トップページ
 研究活動
研究活動
 HPC/AI駆動型医薬プラットフォーム部門紹介
HPC/AI駆動型医薬プラットフォーム部門紹介
 過去のユニット紹介
過去のユニット紹介
 創薬化学AIアプリケーションユニット(本間)
創薬化学AIアプリケーションユニット(本間)
創薬化学AIアプリケーションユニット(本間)
English
ユニットリーダー 本間 光貴
(ほんま てるき) (拠点:横浜地区)
(拠点:横浜地区)- Emailの[at]は@にご変更ください。
- 1993
- 北海道大学大学院理学研究科修士課程修了
- 1993
- 万有製薬株式会社
- 2001
- 北海道大学大学院理学研究科より博士取得(理学博士)
- 2004
- ファイザー株式会社 主幹研究員
- 2007
- 理化学研究所 ゲノム科学総合研究センター 上級研究員
- 2008
- 同 チームリーダー(現職、生命機能科学研究センターに改組)
- 2010
- 同 創薬・医療技術基盤プログラム マネージャー(兼務、現職) 同 創薬分子設計基盤ユニット ユニットリーダー(兼務、現職)
- 2018
- 同 医科学イノベーションハブ推進プログラム 副グループディレクター(兼務) 同 メディシナルケミストリーインテリジェンスユニット ユニットリーダー(兼務)
- 2021
- 同 計算科学研究センター HPC/AI駆動型医薬プラットフォーム部門 副部門長(兼務、現職) 同 創薬化学AIアプリケーションユニット ユニットリーダー(兼務、現職)
キーワード
- インシリコスクリーニング
- ADMET
- FMO 法
- 人工知能
- 深層学習
研究概要
創薬の各段階において、初期ヒットから動物モデルで薬効を示すリードに導く過程(ヒットto リード)は、臨床第二相試験と並ぶボトルネックとなっており、近年の低分子創薬の停滞の原因の一つとなっています。当ユニットは、ヒットto リード及びリード最適化の過程(いわゆる創薬化学プロセス)のAI による効率化を目的としています。創薬プロセスの時間とコストの増加の原因は、活性とADMET(薬物動態と毒性)を同時に最適化することが難しいことです。創薬プロセスを効率的に進めるために、京都大学およびLINC と共同で、新規構造発生、合成経路予測、活性/ ADMET 予測のためのAI モデルを構築しています。
主な研究成果
- 「複数企業の社内データを産学で共有して新薬創出を加速する革新的な枠組みの構築に成功
 」理研プレスリリース(2021年3月8日)
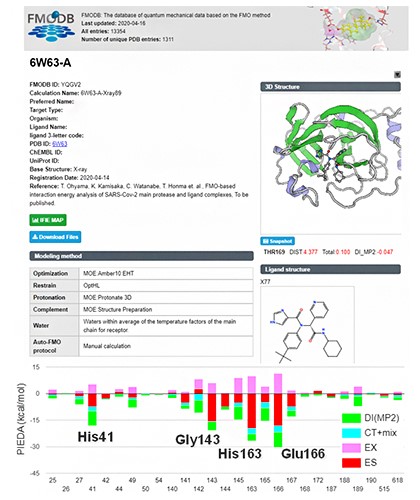
」理研プレスリリース(2021年3月8日) - 「新型コロナウイルス感染症(COVID-19)の治療薬設計に役立つウイルスタンパク質と治療薬候補化合物の相互作用データを公開
 」理研プレスリリース(2020年4月18日、図1)
」理研プレスリリース(2020年4月18日、図1) - 「創薬支援インフォマティクスシステム構築事業成果の商用化について
 」理研プレスリリース(2019年3月18日)
」理研プレスリリース(2019年3月18日) - 「FMO データベース(https://drugdesign.riken.jp/FMODB/)一般公開開始のお知らせ
 」創薬等先端技術支援基盤プラットフォーム(2019 年3 月1 日)
」創薬等先端技術支援基盤プラットフォーム(2019 年3 月1 日) - MEDCHEM NEWS 特集「AI と創薬」 ADMET 予測とそれを活⽤した新規構造提案AI への展開 本間光貴(2018 年11 月号)
- 医薬ジャーナル 特集「人工知能(AI)がもたらす創薬イノベーション」 2)活性・薬効とADMET の同時最適化を可能にする構想提案AI を目指して 本間光貴(2018 年9 月号)
- 心毒性データベースとhERG 予測モデル(https://drugdesign.riken.jp/hERGdb/)
 の公開(2018 年8 月31 日)
の公開(2018 年8 月31 日)
主要論文
- Watanabe C, Okiyama Y, Tanaka S, Fukuzawa K, Honma T.:
“Molecular Recognition of SARS-CoV-2 Spike Glycoprotein: Quantum Chemical Hot Spot and Epitope Analyses”
DOI: 10.1039/D0SC06528E (Edge Article) Chem. Sci., (2021). - Takaya D, Watanabe C, Nagase S, Kamisaka K, Okiyama Y, Moriwaki H, Yuki H, Sato T, Kurita N, Yagi Y, Takagi T, Kawashita N, Takaba K, Ozawa T, Takimoto-Kamimura M, Tanaka S, Fukuzawa K, Honma T.:
“FMODB: The World's First Database of Quantum Mechanical Calculations for Biomacromolecules Based on the Fragment Molecular Orbital Method”
J Chem Inf Model. 22;61(2):777-794. (2021). - Komura H, Watanabe R, Kawashima H, Ohashi R, Kuroda M, Sato T, Honma T, Mizuguchi K.:
“A public-private partnership to enrich the development of in silico predictive models for pharmacokinetic and cardiotoxic properties”
Drug Discov Today. 28:S1359-6446(21)00055-6. (2021) - Kato K, Honma T, Fukuzawa K.:
“Intermolecular interaction among Remdesivir, RNA and RNA-dependent RNA polymerase of SARS-CoV-2 analyzed by fragment molecular orbital calculation”
J Mol Graph Model. 100:107695. (2020). - Kato K, Masuda T, Watanabe C, Miyagawa N, Mizouchi H, Nagase S, Kamisaka K, Oshima K, Ono S, Ueda H, Tokuhisa A, Kanada R, Ohta M, Ikeguchi M, Okuno Y, Fukuzawa K, Honma T.:
“High-precision Atomic Charge Prediction for Protein Systems Using Fragment Molecular Orbital Calculation and Machine Learning”
J Chem Inf Model. 27;60(7):3361-3368. (2020) - Kojima R, Ishida S, Ohta M, Iwata H, Honma T, Okuno Y.:
“kGCN: a graph-based deep learning framework for chemical structures”
Journal of Cheminformatics 12: 1-10. (2020) - Ogura K, Sato T, Yuki H, Honma T.:
“Support Vector Machine model for hERG inhibitory activities based on the integrated hERG database using descriptor selection by NSGA-II”
Scientific reports, 9(1), 1-12. (2019). - Watanabe C, Watanabe H, Okiyama Y, Takaya D, Fukuzawa K, Tanaka S, Honma T.:
“Development of an automated fragment molecular orbital (FMO) calculation protocol toward construction of quantum mechanical calculation database for large biomolecules”
Chem-Bio Informatics Journal, 19, 5-18. (2019). - Yoshikawa N, Terayama K, Honma T, Oono K, Tsuda K.:
“Population-based de novo molecule generation, using grammatical evolution”
Chem Lett, 47(11), 1431-143. (2018). - Sato T, Yuki H, Ogura K, Honma T.:
“Construction of an integrated database for hERG blocking small molecules”
PLoS ONE, 13(7), e0199348. (2018).