トップページ
 研究活動
研究活動
 研究チーム紹介
研究チーム紹介
 計算構造生物学研究チーム
計算構造生物学研究チーム
計算構造生物学研究チーム
English
チームプリンシパル Florence TAMA
(フロハンス タマ) florence.tama[at]riken.jp (拠点:神戸)
florence.tama[at]riken.jp (拠点:神戸)- Emailの[at]は@にご変更ください。
- 2000
- フランス・ポールサバティエ大学計算生物物理学Ph.D.
- 2001
- 米国スクリプス研究所博士研究員
- 2006
- 米国アリゾナ大学助教授
- 2013
- AICS計算構造生物学研究チーム チームリーダー(現職)
- 2015
- 名古屋大学大学院理学研究科教授
キーワード
- モデリング
- 構造
- 機能
- 運動
- 低解像度
研究概要
タンパク質やRNAからなる生体分子複合体は遺伝情報の制御やタンパク質の合成、細胞内輸送などの生命活動に重要な役割を果たしており、それらの異常は様々な疾患を引き起こします。それらの機能を理解して治療につながる薬剤の開発につなげる為には、分子の構造と運動の情報を得ることが重要になります。しかし、これらの複合体は大きいため、幅広く使われている結晶構造解析法を使うことは難しい状況です。そこで、低温電子顕微鏡法やX線小角散乱などの手法が有効になってきます。また新しい技術であるX線自由電子レーザー(XFEL)を構造解析に使う研究も進んでいます。しかし、これらの手法は原子レベルの情報を得ることはできないため、様々な計算機的手法と実験データを組み合わせた、原子モデルを構築する為のアルゴリズムが必要になってきます。当研究室は、「京」や「富岳」などの高性能計算機を利用して実験データから生体分子構造のモデル構築する手法の開発と創薬への応用を目指しています。
主な研究成果
XFEL回折データから3次元構造を再構成
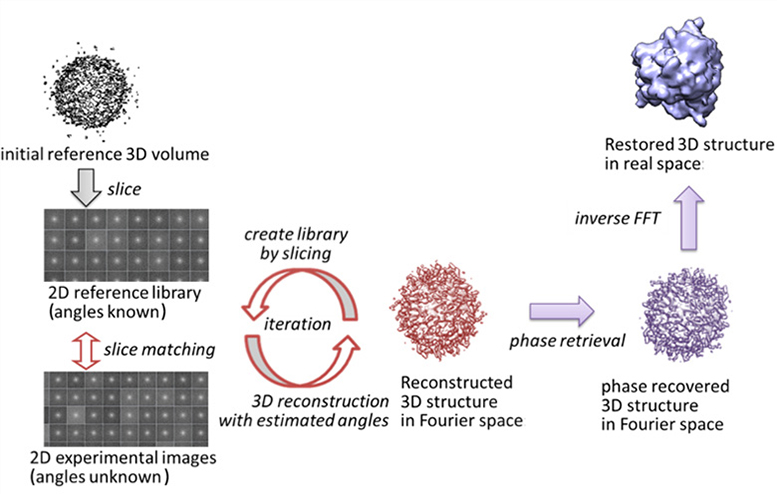
XFELの強力X線源により、生体分子の新しい情報、特に1分子の構造が得られる可能性があります。ただし、現在の実験条件では、X線結晶構造解析法のような1原子解像度はまだ達成されていません。このため、より一般的な利用に向けた実験技術の検討が進む一方で、データ解析のための計算アルゴリズムの開発が行われています。特に、XFELの実験で得られる2次元の回折パターンから、生体分子の3次元構造を再構成するには、レーザービームの入射角を推定するための計算アルゴリズムが必要です。
我々は、単粒子3次元クライオEMの画像生成に広く使われているXMIPPをもとに、XFELのデータ解析のためのプログラムパッケージを開発してきました。XMIPPは実空間の2次元データを扱うように設計されているので、ルーチンの一部をフーリエ空間の2次元データを扱えるように改良しました。反復法により各画像の生体分子の配向を推定し、3次元構造因子振幅を再構成します。また、実空間の3次元モデルは位相回復により得られます。この手法を、ナノ粒子からのXFEL回折パターンに似た実験データに適用することに成功しています。
主要論文
- T. Nagai, F. Tama & O. Miyashita.:
"Cryo-cooling effect on DHFR crystal studied by replica-exchange molecular dynamics simulations"
Biophysical J. 116(3):395-405 (2019). - T. Mori, M. Kulik, O. Miyashita, J. Jung, F. Tama and Y. Sugita.:
"Acceleration of cryo-EM flexible fitting by efficient space partitioning for large biomolecular systems"
Structure 27(1):161-174. (2019). - A. Srivastava, F. Tama, D. Kohda & O. Miyashita.:
"Computational Investigation of the Conformational Dynamics in Tom20-Mitochondrial Presequence Tethered Complexes"
Proteins 87(1):81-90. (2019). - T. Nagai, Y. Mochizuki, Y. Joti, F. Tama & O. Miyashita.:
"Gaussian mixture model for coarse-grained modeling from XFEL"
Optics Express 26(20):26734-26749. (2018). - S. P. Tiwari, F. Tama & O. Miyashita.:
"Searching for 3D structural models from a library of biological shapes using a few 2D experimental images"
BMC Bioinformatics 19:320. (2018). - M. Nakano, O. Miyashita, S. Jonic, A. Tokuhisa, F. Tama.:
"Single-particle XFEL 3D Reconstruction of Ribosome-size Particles based on Fourier Slice Matching: Requirements to Reach Subnanometer Resolution"
J. Synchrotron Rad. 25:1010-1021. (2018). - O. Miyashita, C. Kobayashi, T. Mori, Y. Sugita, F. Tama.:
"Flexible Fitting to Cryo-EM Density Map using Ensemble Molecular Dynamics Simulations"
J. Comp. Chem. 38:1447-1461. (2017). - A. Tokuhisa, S. Jonic, F. Tama and O. Miyashita.:
"Hybrid Approach for Structural Modeling of Biological Systems from X-ray Free Electron Laser Diffraction Patterns"
J. Struct. Biol. 194(3):325-336 (2016). - M. Gallagher-Jones, Y. Bessho, S. Kim, J. Park, S. Kim, D. Nam, C. Kim, Y. Kim, Y. Noh do, O. Miyashita, F. Tama, Y. Joti, T. Kameshima, T. Hatsui, K. Tono, Y. Kohmura, M. Yabashi, SS. Hasnain, T. Ishikawa, C. Song.:
"Macromolecular structures probed by combining single-shot free-electron laser diffraction with synchrotron coherent X-ray imaging"
Nature Commun. 2:5:3798. (2014). -
Q Jin, CO Sorzano, JM de la Rosa-Trevín, JR Bilbao-Castro, R. Núñez-Ramírez, O. Llorca , F. Tama, S. Jonić.:
"Iterative elastic 3D-to-2D alignment method using normal modes for studying structural dynamics of large macromolecular complexes"
Structure 22:496-506. (2014).
Annual Reports
関連プレスリリース等
- 溶液中ナノ粒子を3次元観察できるデータ処理手法 -X線レーザーを用いた生体内に近い環境での構造観察 に期待-
 (2022年7月15日、理化学研究所ウェブサイト)
(2022年7月15日、理化学研究所ウェブサイト)
