トップページ
 研究活動
研究活動
 研究成果 ピックアップ
研究成果 ピックアップ
 新型コロナウイルスのタンパク質のフラグメント分子軌道計算 ~「富岳」による新型コロナウイルス対策その3
新型コロナウイルスのタンパク質のフラグメント分子軌道計算 ~「富岳」による新型コロナウイルス対策その3
新型コロナウイルスのタンパク質のフラグメント分子軌道計算 ~「富岳」による新型コロナウイルス対策その3
新型コロナウイルスのタンパク質のフラグメント分子軌道計算 ~「富岳」による新型コロナウイルス対策その3
立教大学 教授 望月祐志

今回は、立教大学の望月祐志教授らの研究チームによるスーパーコンピュータ「富岳」を使った「新型コロナウイルス関連タンパク質に対するフラグメント分子軌道計算」を紹介します。
これは、新型コロナウイルス表面のスパイクタンパク質やメインプロテアーゼにおける分子同士の相互作用エネルギーを量子力学的に計算することで、タンパク質同士およびタンパク質と薬剤との結合の強さを定量化し、それにより有効な薬剤の開発に役立てようという取り組みです。
(代表者:立教大学 教授 望月祐志)
タンパク質における相互作用の強さを数値化できるFMO法
新型コロナウイルスの表面はスパイクタンパク質と呼ばれる複数の突起で覆われており、このスパイクタンパク質がヒトの細胞の表面にあるACE2受容体というタンパク質と結合することで、新型コロナウイルスはヒトの細胞内に侵入します。また、侵入した新型コロナウイルスはメインプロテアーゼと呼ばれるタンパク質を分解する酵素などを使って我々の体内で増殖します。したがって、これらの働きを薬剤により止めることができれば、ウイルスの侵入および増殖を抑えることができます。
たとえば、スパイクタンパク質とACE2受容体の結合のメカニズムを解明するためのコンピュータシミュレーションの方法の1つとしては、京都大学大の奥野恭史教授の研究チームや理化学研究所の杉田有治チームリーダーの研究チームが進めている「分子動力学計算法(MD法)※1」があります。これは、原子および分子間の相互作用の強さを古典力学であるニュートンの運動方程式を使って計算し、シミュレーションにより可視化するという方法です。
それに対し、本研究チームが行っているのは、「フラグメント分子軌道計算法(FMO法)」と呼ばれるもので、原子および分子間の相互作用の強さを量子力学のシュレディンガー方程式(波動方程式)に基づいて計算し、可視化も交えて解析するという方法です。MD法が原子を古典的な“粒子”として扱っているのに対し、FMO法では電子の“波”としての性質を扱っています(原子核は古典的に扱う近似の下)。
タンパク質の中のアミノ酸同士がどのように相互に作用し合っているか、どのような薬剤分子(化学物質)と強く結合するかといった性質は、量子力学的にはタンパク質を構成するそれぞれのアミノ酸分子の「電子状態」によって決まります。分子の電子状態を解くための方程式がシュレディンガー方程式です。
しかし、シュレディンガー方程式の求解は非常に複雑です。そこで、分子系に対して直截に計算できるように考え出された方法の1つが、「分子軌道法」です。これは、分子を構成している原子の「原子軌道」を足し合わせて「分子軌道」とする計算手法です。そして、さらにこの分子軌道の計算法を、タンパク質などの非常に大きな分子(巨大分子)に適用可能としたのがFMO法です。FMO法は1999年に北浦和夫氏らによって提唱されました。
FMO法では、タンパク質などの巨大分子を「フラグメント」と呼ばれるアミノ酸残基※2 などのより小さな分子の集合体として分割し、それぞれのフラグメントをスーパーコンピュータなどによって並列計算し、最後に統合することで、計算時間やコストの大幅な削減を可能にしています。
FMO法の特徴は、タンパク質の場合、フラグメントとして分割された各アミノ酸残基同士や、タンパク質の活性領域(ファーマコフォア)にはまり込んだ薬剤分子と周囲のアミノ酸残基との間の相互作用エネルギーを、具体的な数値として算出できることにあります。そのため、量子力学的に計算してシミュレーションできるだけでなく、数値を基に相互作用の強さを詳しく解析できるのもFMO法のメリットです。
現在、FMO法に関しては、複数のプログラムが利用可能となっています。その1つが「ABINIT-MP」で、本研究チームの望月祐志教授らによって開発されました。ABINIT-MPの大きな特徴は、フラグメント分割の自動化による使い勝手の良さ、混成並列化によるスーパーコンピュータへの適用性の高さなどです。これまでスーパーコンピュータ「京」などで、ABINIT-MPを用いた新しい創薬基盤の確立に向けた取り組み「FMO創薬コンソーシアム(FMODD)」が進められてきました。
今回、本研究チームはこれまでのFMODDなどでの実績を基に、「富岳」を用いて新型コロナウイルスの侵入と増殖にそれぞれ関与しているスパイクタンパク質とメインプロテアーゼの2つにスポットを当て、ABINIT-MPによる相互作用エネルギーの計算に取り組みました。
ABINIT-MPによるFMO法では、負の電荷によって相互に反発し合いつつ、正の電荷をもつ原子核に引きつけられて運動している電子間の平均的な相互作用からのズレを摂動的に補正することで、計算精度を高めています。現在、摂動補正レベルとしては、電子対間の相関を記述する2次の「MP2」、さらに電子対間の相互作用を考慮した3次の「MP3」などが実装されています。補正レベル(次数)は高ければ高いほど精度は高まりますが、その分、計算の複雑さが急増し、それに伴い処理時間やコストも急増します。
今回、工夫した点は、「富岳」の高い計算能力を2つの方向で活かすことでした。1つは、“同種の計算を大量処理”する「Capacity computing(キャパシティ・コンピューティング)」、もう1つは、“大規模で複雑な問題を計算処理”する「Capability computing(ケイパビリティ・コンピューティング)」です。
- 1 分子動力学計算法(MD法):粒子間の相互作用をクーロンの法則などの物理法則に基づいて計算し、ニュートンの運動方程式を解くことで、分子の動きをシミュレーションする手法。ピコ(1兆分の1)~マイクロ(100万分の1)秒の時間変化を原子レベルの解像度で再現できる。
- 2 アミノ酸残基:アミノ酸同士をつないでいる-CO-NH-結合を「ペプチド結合」と呼ぶ。このように結合しているアミノ酸のことを「アミノ酸残基」という。タンパク質はペプチド鎖を持つ分子のことである。
メインプロテアーゼと薬剤との相互作用の詳細な解析に成功
本研究チームは、まず、Capacity computingの取り組みとして、メインプロテアーゼを対象に、ABINIT-MP によるMP2レベルでのFMO計算を実施しました。
新型コロナウイルスはメインプロテアーゼが、まとめて合成されたタンパク質を目的に合うように分解しながら増殖します。そのため、メインプロテアーゼに結合し、その働きを阻害する新規薬剤の1つとして「N3」と呼ばれる化合物が開発されており、早くも2020年2月には、メインプロテアーゼとN3が結合した複合体の結晶構造がプロテインデータバンク(PDB)※3で公開されています(PDB ID(レコード番号): 6LU7)。
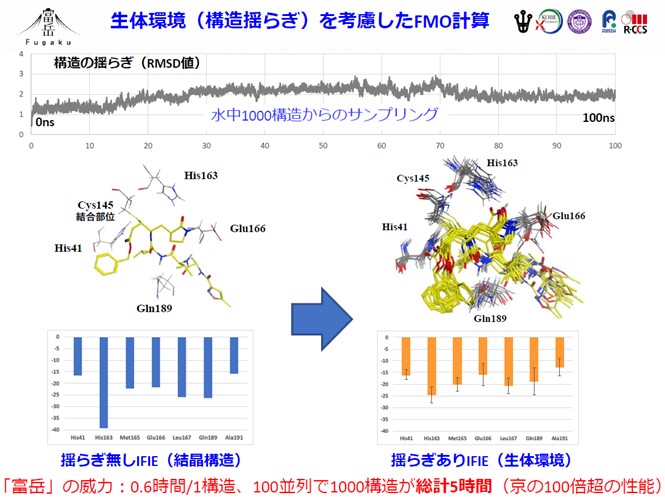
しかし、これはあくまでも静的な結晶構造であり、生体内の動的な環境とは大きく異なります。生体内では絶えず、周囲の水分子などさまざまな分子と相互作用をしており、それにより分子の「構造ゆらぎ」が起こっています。それを考慮しなければ、メインプロテアーゼとN3の相互作用の強さを正しく評価することはできません。
そこで、本研究チームは、まずMD法を使って、1000個におよぶ結晶構造のサンプルを生成し、さらにFMO計算(ABINIT-MPを使用)でこれらの構造サンプルを一括処理しました。このMD法とFMO法の連携により、メインプロテアーゼとN3の動的な相互作用を統計的に評価することにしたのです。
1つの構造サンプルにつき、「富岳」の192ノード(半ラック)を割り当て、並列処理を行った結果、1000個もの構造サンプルをわずか5時間程で完了することができました。小規模なコンピュータであれば数ヶ月はかかる計算です。
また、計算結果からは、従来の静的な結晶構造を基にした解析では、「特定のアミノ酸残基との水素結合が重要な役割を果たしている」という描像が得られていたのに対し、今回のMD法とFMO法の連携による動的な解析では、「複数のアミノ酸残基の水素結合が共同して働いている」という知見が得られました(図1)。この結果から、静的な結晶構造を基にした解析だけでは定量的な評価は難しいことが明らかとなりました。
これは、既存のスーパーコンピュータの計算性能では容易に成し得ない成果です。「富岳」の高い計算性能を活用することで、今後、Capacity computingによる、よりリアルな生体環境を模した動的な解析が可能になることが期待されます。
- 3 プロテインデータバンク(PDB):タンパク質、核酸、糖鎖など生体高分子の3次元構造の原子座標(立体配座)を蓄積している国際的な公共のデータベース。
スパイクタンパク質の構造ゆらぎを計算し構造の安定性を数値化
次に、本研究チームは、 Capability computingの取り組みとして、スパイクタンパク質を対象に、MP3レベルでのABINIT-MPによるFMO計算を実施しました。MP3では、MP2に比して添字を複数持つテンソル量の積和処理が支配的になります。
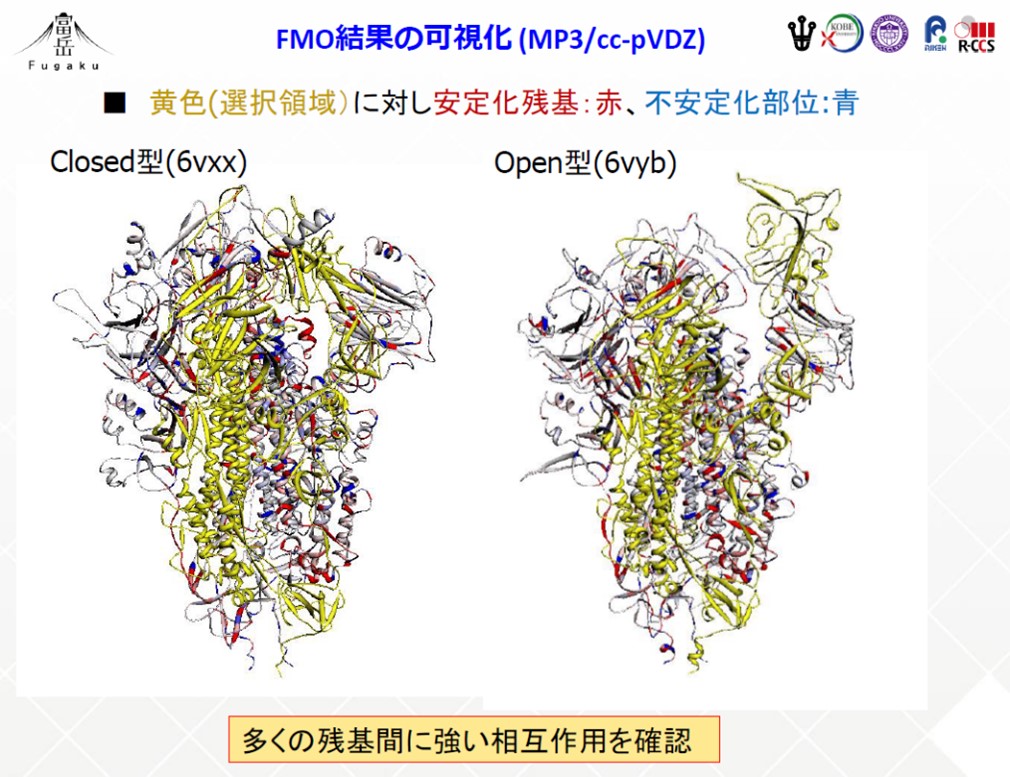
スパイクタンパク質は3つの同じペプチド鎖からなる三量体で、1つの鎖に1100個、合計3300個ものアミノ酸残基で構成された巨大タンパク質です。ヒトの細胞表面のACE2受容体と結合する部分である受容体結合ドメイン(RBD)には、活性型のOpenと不活性型のClosedという2つの立体構造があることがわかっています。Open型は1つのRBDが飛び出したような形をしており、この状態でACE2受容体と結合し、我々の細胞内に侵入します。一方、Closed型は3つのRBDが閉じた形をしています。
そこで、本研究チームは、公開されているOpen型(PDB ID: 6VXX)、Closed型(PDB ID: 6VYB)それぞれの状態のスパイクタンパク質に対してMP3計算を実行しました。
水素原子を含めた総原子数は、5万2千個にもおよびましたが、「富岳」の3072ノード(8ラック)を使って並列処理を行った結果、Closed型構造では3.4時間でMP3計算を、また1.7時間でMP2計算を完了させることができました。これは驚異的な速さと言えます。本来MP3計算の場合、MP2計算に比べて演算量は激増するのですが、「富岳」の計算性能の高さから、わずか2倍のコスト増で、計算を完遂できることがわかりました。
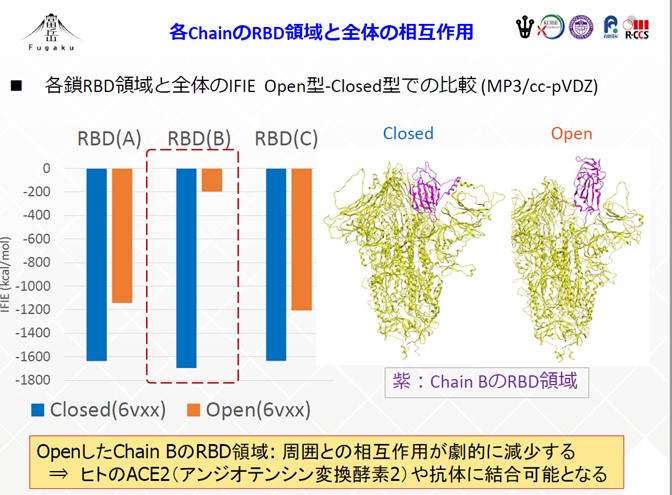
計算結果からは、Open型はClosed型に比べて、安定化エネルギーが低下していること、つまり不安定であるということ(図2)、さらに、Open型のRBDとACE2受容体が結合することで、安定化エネルギーが部分的に回復され得ること、つまり安定性が戻ることが、数値として示されました(ACE2とRBDの結合体(PDB ID: 6M0J)の別計算で確認)。言い換えると、Open型では、3つあるRBDのうちの1つが飛び出したような構造を取ることで、周囲のアミノ酸残基との相互作用が顕著に減少し、代わりにヒトの細胞表面のACE2受容体と結合しやすくなることが数値的に示唆されました(図3)。
加えて、データ科学手法の1つである「特異値分解」を導入することにより、スパイクタンパク質のOpen型とClosed型の構造変化には、電気的に中性のアミノ酸ではなく、アルギニンやグルタミン酸といった荷電しているアミノ酸残基同士の静電的な相互作用が強く働いていることなどもわかってきました。
以上の結果から、従来の「京」では、実用的な時間・コスト内で実現することができなかった構造ゆらぎを含めた統計的なFMO 計算、あるいは演算量の多いMP3レベルでの扱いが、「富岳」を使えば可能となることが明らかとなり、今後、創薬科学や生物物理学における大きなパラダイムシフトが起こる可能性が見えてきました。
また、本研究チームは、イギリスや南アフリカで発見された感染力が強いと言われる新型コロナウイルスの変異型についても計算を行っています。計算結果からは、感染力の強さに符合した数値指標が得られつつあります。
本研究チームは、かつて鳥インフルエンザウイルスのタンパク質などを対象に、FMOによる解析を試みた経験があります(「地球シミュレータ」などを利用)。そうした経験も活かし、今回の新型コロナウイルスの問題に取り組みました。ウイルスとの戦いには終わりがありません。そのため、今後も今回の成果を生かし、新型コロナウイルスだけでなく、他のウイルスに関しても、「富岳」を用いた大規模なFMO計算を実施することで、創薬科学や生物物理学分野への貢献を果たしていきます。
関連リンク
(2021年4月22日)