薬剤耐性化の仕組みを「京」で解く
病原菌に対して薬が効かなくなる薬剤耐性化が、医療の現場で大きな脅威になっています。細菌は、細胞膜にある多剤排出トランスポーターというタンパク質によって、さまざまな薬を細胞外に排出することで、薬を効かなくしてしまうのです。
松永さんは、そのタンパク質のシミュレーションを「京」で行い、わずか1個の水素イオン(プロトン)が付くことでタンパク質全体が変形して薬を排出するという過程を明らかにしました。
運動方程式「F = ma」を究めたい
1997年、神戸大学理学部地球惑星科学科に入学した松永さんは、さまざまな本を読む中で運命の一冊に出会いました。当時、同学科の教授を務めていた伊東敬祐 博士が著した『カオスって何だろう』です。「私はこの本でカオスに興味を持ちました。カオスとは、最初の条件がほんのわずかに違うだけで結果が大きく異なる現象です。私はパソコンでカオスのシミュレーションを行い、思いがけない結果が出るのが面白くて、夢中になりました」
やがて、郡司幸夫 教授・小松崎民樹 准教授の研究室に入り、シミュレーション研究を本格的に学び始めました。「ニュートンの運動方程式 F(力)= m(質量)× a(加速度)を計算することで、太陽系の惑星の軌道からタンパク質の運動まで、さまざまなシミュレーションができます。太陽系のような単純な系でもカオスによる予想外の結果が現れます。F=maの奥深さを知り、私は F=maを究めたいと思いました」
薬剤耐性化を引き起こす多剤排出トランスポーター(AcrB)
博士号を取得した松永さんは、2007年に横浜市立大学の木寺詔紀(あきのり)教授の研究室の研究員に。「そこで、多剤排出トランスポーター(AcrB)のシミュレーションを担当することになりました」
AcrBは、細菌の細胞膜に埋め込まれた膜タンパク質です(図1A)。細菌の中に入り込んださまざまな薬剤を、AcrBが取り込み、細胞外へと排出することで、薬が効かなくなる薬剤耐性化が起きます。
松永さんがAcrBのシミュレーションを担当するまでに、AcrBについてどのようなことが分かっていたのでしょうか。
AcrBなどのタンパク質は、アミノ酸が連なった鎖が立体的に折り畳まれたものです。細胞の中でタンパク質はじっとしているわけではなく、さまざまに構造を変えながら機能を発揮します。そこで、原子間に働く力とF=ma を計算することでその動きを再現します。そのようなシミュレーションを行うには、そのタンパク質がどのような形をしていて、どこにどの種類の原子があるのか、原子スケールの構造情報が必要です。それを知るための代表的な手法が、タンパク質の結晶にX線を当てて解析するX線結晶構造解析です。
AcrBのX線結晶構造解析に成功したのが、村上 聡 教授(現 東京工業大学)たちです。村上教授たちは2002年、大型放射光施設SPring-8により、AcrBのX線結晶構造解析を行い、まったく同じタンパク質が3個合体してできているAcrBの構造を明らかにしました。さらに村上教授たちは2006年、SPring-8により、薬剤がAcrBに結合した状態の構造も明らかにしました。AcrBの3個のタンパク質は少しずつ構造が異なり、それぞれ、薬剤を取り込む構造(取り込み型)、内部に薬剤を結合した構造(結合型)、薬剤を排出する構造(排出型)であることが分かりました。村上教授たちは、3個のタンパク質それぞれが、取り込み型→結合型→排出型と構造が順に変化することで薬剤を排出すると考えました(図1B)。
その構造変化を引き起こす、駆動力は何でしょうか。大腸菌などの細菌は細胞膜の外側に細胞外膜を持ちます。細胞外膜と細胞膜の間は水素イオン(プロトン)の濃度が高い状態で、プロトンは膜タンパク質などを通って、細胞内部へ流れ込みます。AcrBの構造変化は、細胞膜に埋め込まれた下部をプロトンが透過することで引き起こされると考えられます(図1A)。木寺研究室の池口満徳准教授は、AcrBの構造変化に最も影響のあるプロトンが付く場所を明らかにしました。
「では、その場所にプロトンが付くと、どのように構造が変化して薬を排出するのか。そのシミュレーションを私が担当することになったのです。AcrBは、周りの水分子や細胞膜を含めると約50万の原子から成る複雑系です。その原子間に働く力を求め、F=maを計算してAcrBの動きをシミュレーションするには、膨大な計算量が必要です」
2011年に理研 計算科学研究機構に移った松永さんは、計算手法を工夫し、「京」を使ってAcrBのシミュレーションを行うことを目指しました。そして、プロトンが付くことでAcrBが結合型から排出型へ構造を変化させて薬剤を排出する過程をシミュレーションすることに成功しました(図1C)。
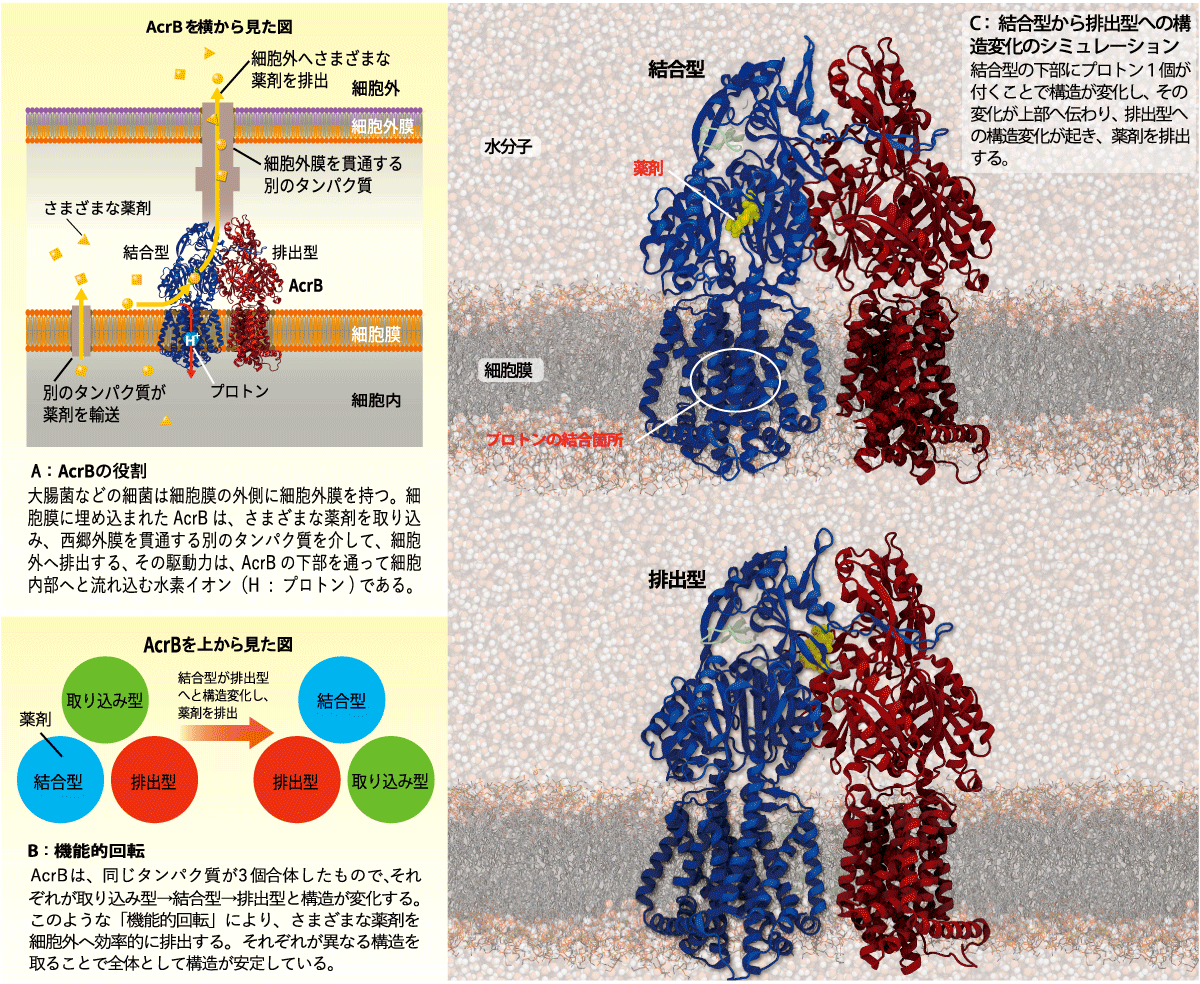

大腸菌などの細菌は細胞膜の外側に細胞外膜を持つ。細胞膜に埋め込まれたAcrBは、さまざまな薬剤を取り込み、細胞外膜を貫通する別のタンパク質を介して、細胞外へ排出する。その駆動力は、AcrBの下部を通って細胞内部へと流れ込む水素イオン(H+:プロトン)である。
B:機能的回転
AcrBは、同じタンパク質が3個合体したもので、それぞれが取り込み型→結合型→排出型と構造が変化する。このような「機能的回転」により、さまざまな薬剤を細胞外へ効率的に排出する。それぞれが異なる構造を取ることで全体として構造が安定している。
C:結合型から排出型への構造変化のシミュレーション
結合型の下部にプロトン1個が付くことで構造が変化し、その変化が上部へ伝わり、排出型への構造変化が起き、薬剤を排出する。
足元に付いたアリがゾウの全身を動かす謎
「薬剤を取り込み排出するのはAcrBの上部で、プロトンが付く下部とは遠く離れています。50万もの原子から成る巨大なシステムにプロトンが1個付くだけで、結合型から排出型へという大きな構造変化がなぜ起きるのか、それが大きな謎です。ものすごく大げさに言うと、ゾウの足元に付いた1匹のアリが、ゾウの全身を動かすようなものです」
AcrBのシミュレーションにより、予想外のことが分かりました。「プロトンが付かない状態では、AcrBの結合型にはプラスやマイナスの静電ポテンシャルの偏りがほとんどありません。ところが、プロトンが1個付くだけで、プラスに偏った領域が広がり、大きく不安定化します。プロトン1個により、これほど大きく不安定化することに驚きました」(図2)
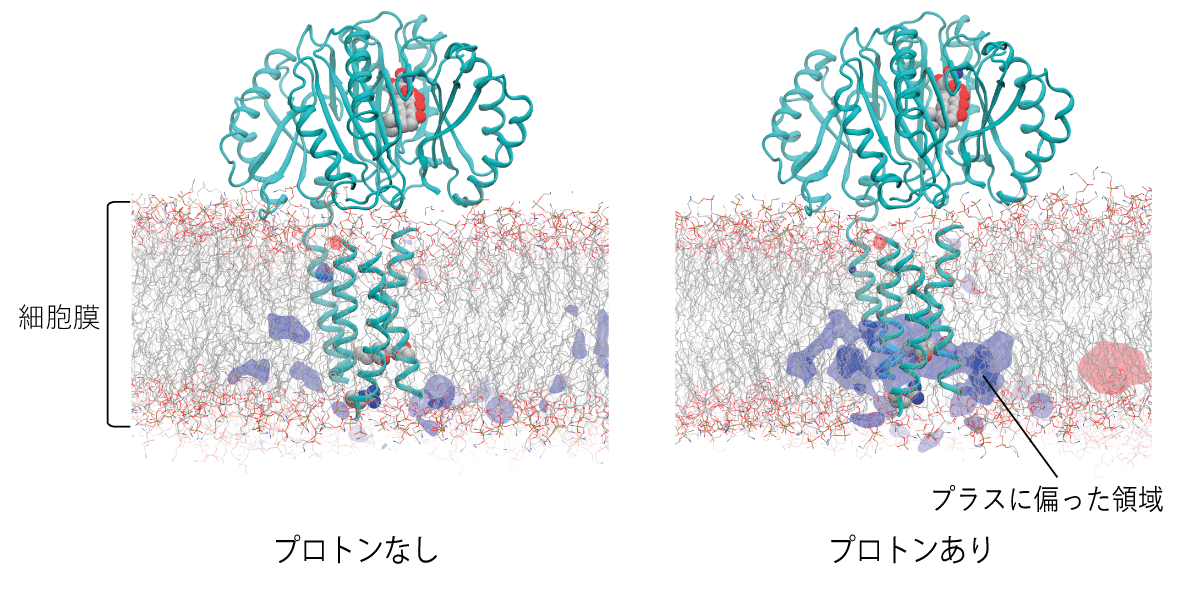

そのプラスに偏った領域を解消するように、AcrBの下部の構造が変化し、その変化が上部へ伝わり、排出型に構造変化することが分かりました。「その構造変化には、下部にあるいくつかのアミノ酸が重要なことが分かっています。しかし、これほど大きく不安定化するには、重要な仕組みがほかにも隠されているはずです。結合型でプロトン1個が付いていない状態と付いた状態を比較することで、その仕組みを解明していきたいと思います」
アロステリーの共通原理を探る
病気の原因となるタンパク質に結合して、その働きを阻害する分子が薬となります。「AcrBの場合、薬剤を結合する上部だけでなく、プロトンが付く下部の変化を上部へ伝える連結部の動きを止めることでも、薬剤排出機能を阻害することができるのではないかと考えています」
タンパク質の機能する仕組みが分かることで、それを阻害する薬の標的箇所の選択肢が広がるのです。従来、阻害することが難しかったタンパク質に対しても、別の箇所を標的にすることで薬をつくることができるようになると期待できます。
「ある場所のわずかな変化が、遠く離れたほかの場所の変化を引き起こすことを、アロステリーと呼びます。AcrBが機能する仕組みもアロステリーの例として興味深いと思っています」
さまざまなタンパク質がアロステリーによって機能を発揮します(図3)。「私は、アロステリーには共通の原理があると考えています。さまざまなタンパク質のアロステリーをシミュレーションで再現して、そこから共通原理を探りたいと考えています。そのために、将来的にはシミュレーションデータから似た特徴を見つけ出す機械学習などの技術を利用する必要があるかもしれません」

アロステリーの共通原理が分かれば、創薬にも大いに役立つはずです。
生命科学の新時代が到来
たくさんの原子から成るタンパク質を計算するには膨大な計算量が必要なため、従来のシミュレーションで動きを再現できる時間はマイクロ(100万分の1)秒くらいまででした。松永さんのAcrBのシミュレーションは、結合型から排出型へのミリ(1000分の1)秒に相当する構造変化を再現することに成功しています。
「シミュレーションと実験の結果を直接比較して生命現象を深く理解することが、ようやく可能になってきました。シミュレーションによってタンパク質のある箇所が機能に重要であることが予測できれば、その箇所を改変することで機能がどう変わるか調べて予測を検証する実験ができます」
実験では、タンパク質の構造変化が起きる前と後を観察することはできても、変化の最中を観察することは難しいという課題があります。「AcrBでも結合型にプロトン1個が付いて不安定化した状態は一瞬なので、観察することはとても難しいでしょう。シミュレーションならば、タンパク質の機能にとって重要な瞬間を原子スケールで見て、生命現象の仕組みを物理の言葉で説明することができます」
シミュレーションが、実験と並ぶ主要な手法となる生命科学の新時代が始まっています。
(取材・執筆:立山 晃/フォトンクリエイト)
に収録されています。
